Mon 1er coup de cœur pour R, je l’ai eu en découvrant les 2 fonctions de transposition pivot_wider et pivot_longer. Remettre une table au carré (je devrais dire au rectangle) est un besoin tellement fréquent pour qui veut produire un graphique ou une carte à partir d’un jeu de données récupéré sur le web !
Jadis (?) on faisait des tableaux croisés dynamiques avec son tableur, pour bien exposer à l’écran les relations entre deux notions, l’une déployée en ligne, l’autre en colonne. Les statisticiens ont l’habitude aussi des tables de contingence, qui visualisent la dépendance entre deux caractères sous forme de comptages, de sommes et/ou de pourcentages.
Quand on génère un tableau croisé, on passe d’une structure « longue » à une structure « large« . La structure longue a beaucoup de lignes et peu de colonnes. La structure large équilibre mieux les deux dimensions, elle est plus lisible. C’est aussi celle que l’on retrouve dans un graphique, car elle exploite au mieux les deux dimensions du plan.
Mais pour transformer un jeu de données avant sa représentation finale, la structure longue est la plus pratique, car les opérations statistiques portent avant tout sur des colonnes (filtrages, agrégations, calcul de nouveaux indicateurs…) Chaque colonne dans une table statistique bien construite (« tidy » en terminologie R/Wickham) a un contenu homogène, et les colonnes différencient des axes et des indicateurs, en nombre bien maitrisé.
Une série de traitements se conduit mieux avec des structures longues et ne produit du « large » qu’en bout de chaine, quand il s’agit d’éditer un tableau d’analyse ou une datavisualisation. C’est aussi pourquoi les données sont généralement stockées en base sous une forme longue. Enfin, les outils de datavisualisation en ligne promeuvent de plus en plus le format long en entrée.
Mais ce que l’on trouve en OpenData est assez souvent en format « large« , quand le producteur a privilégié la lisibilité de l’exposition à la facilité du remodelage. Ces deux critères sont également respectables. Avec la boîte à outils R, pas de souci, nous sommes vraiment armés pour le tout terrain !
Je vais m’appuyer dans cet article sur des exemples concrets, qui vous parleront je l’espère plus facilement que les exposés habituels sur les jeux iris ou mtcars fournis par défaut dans R.
Je vais vous montrer, en utilisant les options les plus avancées des fonctions de pivot et exploitant des données OpenData Insee :
- comment produire du format long à partir du large,
- et réciproquement du large à partir du long, même si le besoin est moins fréquent,
- et enfin comment simplement transposer une table, en échangeant 2 critères qualitatifs, sans forcément qu’elle ne devienne radicalement plus longue ou plus large.
1 – Naissances millésimées : isoler l’année dans une nouvelle colonne
Nous reprenons la base Insee historique des naissances domiciliées présentée dans cet article :
library(tidyverse); library(curl); library(readxl); library(janitor)
url_ec <- "https://www.insee.fr/fr/statistiques/fichier/1893255/base_naissances_2019.zip"
xlsfile <- str_replace(basename(url_ec), ".zip", ".xlsx") # "base_naissances_2019.xlsx"
curl_download(url_ec, tempzip <- tempfile()) # télécharge le zip
xlsfile <- unzip(zipfile = tempzip, files = xlsfile) # extrait le xlsx
tb_com <- read_excel(xlsfile, sheet = 1, skip = 5) %>%
clean_names() %>% select(-(libgeo:dep))
# clean_names de janitor normalise les colonnes => minuscules sans blancs ni accents
tb_com_tr <- tb_com %>%
pivot_longer(cols = starts_with('naisd'),
names_to = "annee", values_to = "naisd")
# par défaut, la nouvelle variable annee reprend les noms de colonnes numériques
# il nous faut la corriger en remplaçant la racine 'naisd' par '20', ainsi 'naisd12' => '2012'
tb_com_tr <- tb_com_tr %>%
mutate(annee = str_replace(annee, "naisd", "20"))
Voilà qui nous amène à une table de 3 colonnes et 350 000 lignes (ici les 20 premières) :
tb_com_tr <- tb_com %>%
pivot_longer(cols = starts_with('naisd'), names_to = "annee", values_to = "naisd",
names_prefix = "naisd") # cette chaîne est soustraite, 'naisd12' => '12'
tb_com_tr <- tb_com %>%
pivot_longer(cols = starts_with('naisd'), names_to = "annee", values_to = "naisd",
names_pattern = "naisd(\\d+)") # extraction du contenu entre ()
# il nous faut préfixer annee par "20", ce qui est possible avec names_transform
tb_com_tr <- tb_com %>%
pivot_longer(cols = starts_with('naisd'), names_to = "annee", values_to = "naisd",
names_pattern = "naisd(\\d+)", names_transform = list(annee = ~str_c('20',.x)))
# combinaison extraction des chiffres après naisd et fonction de concaténation
tb_com_tr <- tb_com %>%
pivot_longer(cols = starts_with('naisd'), names_to = "annee", values_to = "naisd",
names_transform = list(annee = ~str_c('20', str_sub(.x, 6))))
# une seule fonction pour fabriquer la colonne annee
Dans cet exemple, nous avons fait passer une collection de variables (en ligne) par transposition vers 2 nouvelles colonnes : 1 axe qualitatif temporel et 1 indicateur numérique unique. Confier tous les traitements à la seule fonction pivot_longer est un gage de rapidité, car elle sait mettre en œuvre des optimisations de bas niveau.
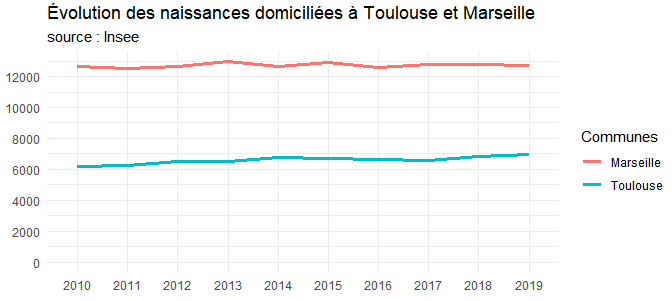
ggplot(tb_com_tr %>% filter(codgeo %in% c('31555','13055')) ) +
aes(x = annee, y = naisd, colour = codgeo, group = codgeo) +
geom_line(size = 1.2) +
labs(title = "Évolution des naissances domiciliées à Toulouse et Marseille",
color = "Dépts", subtitle="source : Insee", x = "", y = "" ) +
theme_minimal() +
expand_limits(y = 0) +
scale_y_continuous(breaks = seq(0, 14000, 2000)) +
scale_color_discrete(name = "Communes", labels = c("Marseille", "Toulouse"))
2 – Types de familles : dégager 2 nouveaux axes qualitatifs
Voyons dans ce nouvel exemple comment dégager deux axes qualitatifs au lieu d’un seul. Je m’appuie ici sur un tableau détaillé du recensement, millésimé 2017, accessible à partir de cette page Insee. Il s’agit de données communales par type de famille et nombre d’enfants de moins de 25 ans.
zip_url <- "https://www.insee.fr/fr/statistiques/fichier/4515481/BTX_TD_FAM1_2017.zip" xlsfile <- unzip(curl_download(zip_url, tempfile(), quiet = F)) tb_com <- read_excel(xlsfile, sheet = 1, skip = 10) %>% select(-LIBGEO)
# A tibble: 34,951 x 61 CODGEO NBENFFR0_TF1211 NBENFFR0_TF1212 NBENFFR0_TF1221 NBENFFR0_TF1222 NBENFFR0_TF1231 1 01001 0 0 0 0 30.6 2 01002 0 0 0 0 15.3 3 01004 3.17 15.8 14.5 48.0 484. 4 01005 0 0 5.30 10.3 79.4 5 01006 0 0 0 0 10.1 6 01007 0 0 0 0 112. 7 01008 0 0 0 4.98 34.9 8 01009 0 0 0 5.09 20.4 9 01010 0 0 0 5 30 10 01011 0 0 0 0 0
La doc du fichier précise que ce tableau présente des comptages issus du croisement de 2 critères qualitatifs :
- NBENFFR : nb. d’enfants, modalité codée de ‘0’ à ‘4’
- TF12 : type de famille, modalité codée de ’11’ à ’44’
L’information peut se lire dans les noms de colonnes du fichier.
Ainsi NBENFFR2_TF1244 correspond à la définition NBENFFR = ‘2’ et TF12 = ’44’ (couple sans emploi avec 2 enfants de moins de 25 ans).
tb_com_tr <- tb_com %>%
pivot_longer(!CODGEO, names_to = c("NBENFFR","TF12"), values_to = "NB",
names_pattern = "NBENFFR(\\d+)_TF12(\\d+)")
# !CODGEO cible toutes les variables sauf CODGEO
# \\d cible un chiffre (digit), équivalent à [0-9] ou [:digit:]
# \\d+ cible une répétition quelconque de chiffres
# l'écriture ci-dessus fonctionne mais est paresseuse,
# le + spécifiant un nb. quelconque de chiffres
# une "regexp" plus stricte s'écrirait :
tb_com_tr <- tb_com %>%
pivot_longer(!CODGEO, names_to = c("NBENFFR","TF12"), values_to = "NB",
names_pattern = "NBENFFR([0-9]{1})_TF12([0-9]{2})")
Voici les données dans la table transposée tb_com_tr (ici les 20 premières lignes) :
Comme on le voit une telle structure est bien plus facile à manipuler. Que l’on songe par exemple à la sélection suivante : familles monoparentales de plus de 4 enfants (TF12 <= ’22’ && NBENFFR = ‘4’). Dans la structure large initiale, il aurait fallu identifier et lister méticuleusement les colonnes concernées.
tb_com_fl <- tb_com_tr %>%
filter(TF12 <= '22' & NBENFFR == '4') %>%
pivot_wider(names_from = c("NBENFFR","TF12"), values_from = "NB",
names_glue = "NBENFFR{NBENFFR}_TF12{TF12}")
# names_glue permet des substitutions dynamiques au sein des {}
Pivot_wider présente une syntaxe tout à fait symétrique de celle de pivot_longer. Elle fonctionne d’autant mieux que le jeu à transposer vers un format large dispose d’une clé primaire (ici CODGEO) qui identifie bien chaque enregistrement de façon unique. Si tel n’était pas le cas (vous travaillez par exemple sur un jeu d’observations élémentaires), et qu’un warning apparaissait, ajouter un incrément automatique de type fid = row_number() ou cumsum(…) vous tirera d’affaire.
3 – Chiffres clés depuis 1968 : transposer 2 groupes de variables, naissances et décès
Poursuivons notre exploration de pivot_longer. Jusqu’à présent, nous avons dans le passage à la structure longue réduit les colonnes numériques initiales à une seule, car nous n’avions qu’un indicateur à considérer (naissances ou nombre de familles). Voyons comment gérer le cas de deux indicateurs (ou plus), avec une base de départ qui comprend des informations plus variées sur les naissances, les décès, les logements, la population, etc.
urlcc <- str_c("https://www.insee.fr/fr/statistiques/fichier/4171585/",
"base-cc-serie-histo-2016-xls.zip")
xlsfile <- unzip(curl_download(urlcc, tempfile()))
tb_cc <- read_excel(xlsfile, sheet = 1, skip = 5) %>% clean_names()
tb_cc <- tb_cc %>% select(codgeo, starts_with("nais"), starts_with("dece"))
# A tibble: 34,953 x 15
codgeo nais1116 nais0611 nais9906 nais9099 nais8290 nais7582 nais6875 dece1116 dece0611
1 01001 41 48 67 60 47 28 37 25 29
2 01002 21 13 26 20 10 4 5 7 9
3 01004 1114 1005 1158 1299 1323 1161 1220 595 503
4 01005 101 114 116 135 86 69 99 42 43
5 01006 9 4 6 6 3 2 5 6 5
6 01007 163 156 199 198 159 151 128 69 67
7 01008 55 53 44 59 62 23 27 19 16
8 01009 10 26 18 21 17 11 10 11 16
9 01010 57 72 70 70 63 67 48 30 35
10 01011 21 24 34 39 36 26 40 9 7
tb_cc_tr <- tb_cc %>%
pivot_longer(!codgeo, values_to = "nb", names_to = c("stat","periode"),
names_pattern = "([a-z]+)(\\d+)" )
Voici la nouvelle table pivotée (10 premières lignes) :
# fonction de recodage de la période '8290' => '1982-1990', '0611' => '2006-2011'
f <- function(periode) {
p1 = str_sub(periode,1,2)
p2 = str_sub(periode,3,5)
p1 = ifelse( p1 > '3', str_c('19', p1), str_c('20', p1) )
p2 = ifelse( p2 > '3', str_c('19', p2), str_c('20', p2) )
str_c(p1, "-", p2)
}
# transposition avec recodage de la période
tb_cc_tr <- tb_cc %>%
pivot_longer(!codgeo, values_to = "nb", names_to = c("stat","periode"),
names_transform = list(periode = f),
names_pattern = "([a-z]+)(\\d+)" )
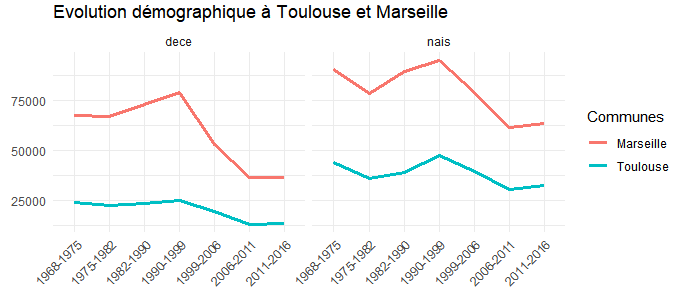
# mise en graphique en exploitant les 'facet', démultipliant les visualisations
# pour avoir une courbe des décès à côté de la courbe des naissances
ggplot(tb_cc_tr %>% filter(codgeo %in% c('31555','13055')) %>% arrange(periode),
aes(x = periode, y = nb, group = codgeo, colour = codgeo)) +
facet_wrap(~stat) +
geom_line(size = 1.2) +
theme_minimal() +
theme(axis.text.x = element_text(angle = 45, hjust = 1)) +
labs(title = "Evolution démographique à Toulouse et Marseille", x = "", y = "") +
scale_color_discrete(name = "Communes", labels = c("Marseille", "Toulouse"))
tb_cc_tr <- tb_cc_tr %>%
pivot_wider(names_from = 'stat', values_from = 'nb')
Ce qui me donne :
Avec cette structure, je vais pouvoir calculer plus facilement un solde naturel comme nais-dece.
4 - Utilisation avancée de pivot_longer
tb_cc_tr <- tb_cc %>%
pivot_longer(!codgeo, values_to = "nb", names_to = c(".value", "periode"),
names_pattern = "([a-z]+)(\\d+)")
# le mot clé .value désigne les variables extraites (nais/dece) à ne pas transposer
# c'est-à-dire à conserver comme colonnes
# tb_cc : table d'origine
codgeo nais1116 nais0611 nais9906 nais9099 nais8290 nais7582 nais6875 dece1116 dece0611
1 01001 41 48 67 60 47 28 37 25 29
2 01002 21 13 26 20 10 4 5 7 9
3 01004 1114 1005 1158 1299 1323 1161 1220 595 503
4 01005 101 114 116 135 86 69 99 42 43
# tb_cc_tr : table transposée
codgeo periode nais dece
1 01001 1116 41 25
2 01001 0611 48 29
3 01001 9906 67 36
4 01001 9099 60 45
5 01001 8290 47 30
6 01001 7582 28 31
7 01001 6875 37 25
8 01002 1116 21 7
9 01002 0611 13 9
10 01002 9906 26 16
spec <- tb_cc %>%
build_longer_spec(!codgeo, values_to = "nb", names_to = c(".value", "periode"),
names_pattern = "([a-z]+)(\\d+)")
# spec : A tibble: 14 x 3
.name .value periode
1 nais1116 nais 1116
2 nais0611 nais 0611
3 nais9906 nais 9906
4 nais9099 nais 9099
5 nais8290 nais 8290
6 nais7582 nais 7582
7 nais6875 nais 6875
8 dece1116 dece 1116
9 dece0611 dece 0611
10 dece9906 dece 9906
11 dece9099 dece 9099
12 dece8290 dece 8290
13 dece7582 dece 7582
14 dece6875 dece 6875
tb_cc_tr <- tb_cc %>% pivot_longer_spec(spec) # tb_cc_tr : table transposée codgeo periode nais dece 1 01001 1116 41 25 2 01001 0611 48 29 3 01001 9906 67 36 4 01001 9099 60 45 5 01001 8290 47 30
build_longer_spec => crée une table spec => pivot_longer_spec => crée la table transposée
Du coup, si l’on comprend bien la logique de cette table de spécifications, il est possible de l’aménager à son gré pour définir des transpositions qui ne seraient pas possibles avec les paramètres actuels de pivot_longer (fonction qui est une sorte d’assistant pour définir une spéc). Je vais à titre d’exemple affiner la table spec précédente et la faire exécuter.
# fonction de recodage '06'=>'2006', '75'=>'1975'
f2 <- function(x) str_c(ifelse(str_sub(x,1,1) == '0','20','19'), x)
# modification de la spécification de transposition
# recodage de la période avec f2 et renommage des variables nais et dece
spec2 <- spec %>%
mutate(`.value` = recode(`.value`, 'nais' = 'naissances','dece' = 'deces')) %>%
separate(periode, into = c("a1","a2"), sep = 2) %>%
mutate(a1 = f2(a1), a2 = f2(a2)) %>%
unite("periode", a1:a2, sep = '-')
# spec2 : A tibble: 14 x 3
.name .value periode
1 nais1116 naissances 1911-1916
2 nais0611 naissances 2006-1911
3 nais9906 naissances 1999-2006
4 nais9099 naissances 1990-1999
5 nais8290 naissances 1982-1990
6 nais7582 naissances 1975-1982
7 nais6875 naissances 1968-1975
8 dece1116 deces 1911-1916
9 dece0611 deces 2006-1911
10 dece9906 deces 1999-2006
11 dece9099 deces 1990-1999
12 dece8290 deces 1982-1990
13 dece7582 deces 1975-1982
14 dece6875 deces 1968-1975
# application de la nouvelle spec2
tb_cc_tr <- tb_cc %>% pivot_longer_spec(spec2)
# tb_cc_tr : A tibble: 244,671 x 4
codgeo periode naissances deces
1 01001 1911-1916 41 25
2 01001 2006-1911 48 29
3 01001 1999-2006 67 36
4 01001 1990-1999 60 45
5 01001 1982-1990 47 30
6 01001 1975-1982 28 31
7 01001 1968-1975 37 25
8 01002 1911-1916 21 7
9 01002 2006-1911 13 9
10 01002 1999-2006 26 16
5 – Données par pays de la Banque mondiale
J’ai travaillé récemment sur des indicateurs de santé en Amérique du Sud, tirés de la base de données de la Banque mondiale.
tb_wb <- world_bank_pop %>% select(-(`2000`:`2009`))
Comme on le voit, après avoir un peu allégé cette table pour la lisibilité, les années sont présentées en colonnes, mais on a cette fois-ci un axe « indicator » qui décline toute une série d’indicateurs par pays. Cette structure n’est à l’évidence pas très maniable.
tb_wb_tr <- tb_wb %>% pivot_longer(`2010`:ncol(.), names_to = "annee", values_to = "value") %>% pivot_wider(names_from = "indicator", values_from = "value") %>% clean_names() # normalise les noms de colonne
Récapitulatif
Les fonctions de pivot de R sont puissantes et rapides, mais demandent à être apprivoisées (se familiariser avec les expressions régulières est un avantage certain). Elles permettent de remettre en forme pratiquement tout jeu de données, afin d’en faciliter le maniement et l’analyse ultérieure. pivot_longer et pivot_wider sont au cœur de la philosophie élégante et fluide de R/Tidyverse. Prenant la suite de gather et spread, elles continuent d’évoluer avec des options introduites pour résoudre des problèmes très pratiques.
Pour aller plus loin
La documentation de référence (en anglais), et une présentation du concepteur :
Des explorations voisines (en français) :
Merci pour ce tuto. Pour le moment j’utilisais plutôt gather/spread. Je vais me pencher sur les fonctions de pivot qui semblent très puissantes dans ces exemples.
Une remarque sur l’exemple 2 du TD FAM de l’Insee, ça laisse songeur de savoir que les données sont au départ exactement sous la forme « longue », qu’elles sont retraitées à l’Insee pour être diffusées en version « large » et finalement remise en version « longue » par les utilisateurs. Peut être faudrait il laisser les données à l’état initial et laisser les utilisateurs décider de la structure qu’ils souhaitent. C’est d’ailleurs exactement ce qui est fait proposé via les API Insee (notamment l’API DDL), mais c’est probablement réservé à un public expert.
Merci Régis, je comprends votre interrogation quant au format idéal de diffusion. L’idéal c’est qu’il y ait les deux, car le format ‘long’ peut dérouter les non-habitués, alors que le large a aussi ses avantages (meilleur exposé des variables, vision immédiate de toutes les données d’une commune, etc.) Le chemin des API complémentaires au mode de diffusion actuel me parait une bonne réponse, dès lors bien sûr que l’API est simple et rapide d’usage.
pivot_longer et pivot_wider remplacent gather et spread, avec selon leur concepteur H. Wickham le souhait d’une dénomination plus facile à retenir, et des ajouts fonctionnels en partie empruntés à l’univers de l’excellent data.table.